天津大学DNA存储团队提出DNA存储基于隐藏参考序列的读出新方法
2026-01-21
本站讯(通讯员:刘双)随着全球数据量的爆炸式增长,传统存储方式的能耗与成本都面临巨大的挑战。DNA数据存储凭借存储密度高、可用时间久、维护成本低等特点,已成为应对海量数据长期存储的重要解决途径。同时,DNA存储融合了信息、半导体、生物等诸多领域,是碳硅融合发展的重要交叉方向。《半导体十年计划》(ISA&SRC, 2021)、《国际器件与系统路线图(大规模数据存储)》(IEEE, 2023)都将DNA存储作为未来大规模数据存储的主要介质之一。《中华人民共和国第十四个五年规划与2035年远景目标纲要》也将DNA存储与量子计算、量子通信、神经芯片并列,作为重点布局的四项前沿技术之一。然而,DNA存储在实际应用中仍存在错误类型复杂、数据处理复杂度高的问题,这已成为制约其大规模应用的重要瓶颈。
近日,微电子学院陈为刚教授团队与合成生物技术全国重点实验室元英进院士团队联合,在长片段DNA存储的低测序深度可靠读出方面取得进展。研究团队提出了一种DNA存储快速“自启动式”可靠读出方案,通过构建多重隐藏参考序列,将DNA存储读出从未知物种“从头测序”问题转变为 “重测序”问题,可有效区分并逐步利用不同错误特点的测序数据,实现极低测序深度的读出,降低了DNA存储读出成本与复杂度。研究成果于近日发表在国际重要期刊iMeta(影响因子33.2),论文题目“Fast bootstrap and reliable readout using hidden references for DNA data storage”(论文链接:https://doi.org/10.1002/imt2.70105)。
大片段DNA存储数据可充分利用细胞内组装与复制等优势,在大规模数据复制与分发领域有潜在应用。大片段DNA存储的数据读出与未知物种的基因组从头测序非常类似。传统方法通常依赖测序数据重叠区域比对,测序深度要求高;数据需要全局处理、计算复杂度非常高。针对上述问题,本项研究面向叠加水印编码的大片段DNA,从简单到复杂,依次构建三种隐藏参考序列,包括嵌入已知序列、初步组装的骨架参考序列、迭代译码恢复的全局参考序列,分阶段识别具有不同错误特点的测序数据(称为读段),实现了“从头测序”到 “重测序”问题的转化;进一步,利用基于隐马尔科夫过程的机器学习算法对读段发生的插入与删除(insertion/deletion,Indel)错误就行纠正,实现了大片段DNA存储的快速“自启动式”可靠读出,需要的测序深度远低于组装策略(图1)。本研究的主要创新包括:
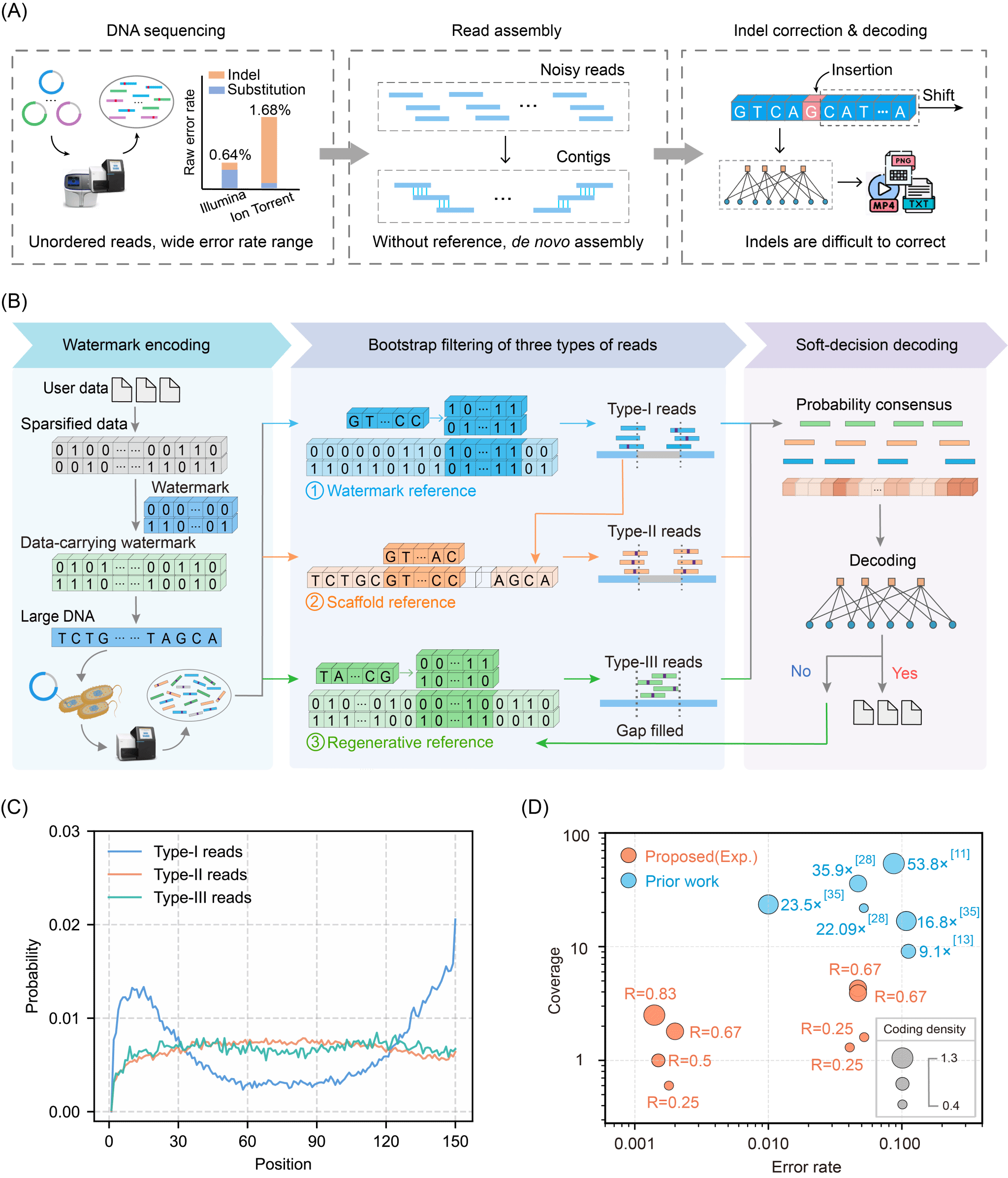
图1 基于多重隐藏参考的大片段DNA数据存储读出方法
第一,大片段DNA数据读出中,针对传统从头组装方法需较高测序数据深度和计算复杂度等问题,通过构建多重隐藏参考序列,将从头读出转化为类似重测序的工作流程,实现对不同错误特性读段的分阶段识别与高效纠错利用。
第二,针对仅发生替换错误或者indel错误在两端的测序读段,利用序列构建时叠加的水印序列作为隐藏参考,通过测序读段与参考序列滑动相关实现读段定位,并利用逐比特概率合并生成软判决信息,实现快速译码恢复。为适应不同错误率,研究设计了编码码率为1/4、1/2、2/3、5/6阶梯码率的低密度奇偶校验(LDPC)码用于匹配不同的错误与测序深度。
第三,针对Indel错误发生在序列中间位置的测序读段,首先利用相关成功读段构建骨架参考序列作为第二轮参考序列,进行比对识别,然后利用机器学习算法纠正每一条测序读段的Indel错误;进一步,将LDPC码基于因子图迭代译码结果重新生成参考序列,称为再生参考序列,针对覆盖度较低且包含Indel错误的读段,进行新一轮比对以填补低覆盖度区域,实现可靠数据恢复。
第四,该方法可兼容不同错误特性的DNA测序技术体制(例如二代Illumina测序、三代纳米孔测序),针对不同DNA的实测数据都实现了低覆盖度下的无错恢复。针对出错率非常高的纳米孔测序实测数据,提出的方法通过将测序读段进行规则分段,仍可以利用提出的高效识别方法,无需复杂的组装与磨光过程,降低了大片段DNA基于纳米孔测序读出的处理复杂度。
本项研究进一步拓展了叠加编码潜力,提出了DNA存储测序数据直接定位处理方法,结合基于机器学习算法的测序数据纠错方法,有效降低DNA存储读出的成本与复杂度。
天津大学DNA存储研究团队以元英进院士为学术带头人,依托合成生物技术全国重点实验室、教育部前沿科学研究中心等,开展了合成DNA用于数据存储的深度交叉研究。微电子学院陈为刚教授团队自2011年以来,从事纠正符号插入与删除(Insertion/Deletion)错误的编码研究,在国家自然科学基金、教育部新世纪优秀人才资助计划、国家重点研发计划等项目的支持下,提出了纠正Indel错误的复合信号设计方法与迭代处理的系列化方法。团队从2017年起开展该类纠错编码与DNA存储的融合研究,提出了基于叠加编码的酵母人工染色体数据存储新模式(写入成本高,便于复制与读出,简称“光盘模式”)。目前,在中长片段DNA数据存储方面,研究团队已形成了系列化的信息可靠编码与数据读出方法(National Science Review, 2021; Briefings in Bioinformatics, 2025; Nature Communications, 2025; iMeta, 2026)。相关研究拓展了可应用于数据存储的合成DNA介质类型,为未来构建DNA存储专用器件与装备提供支撑。
2026-01-21
天津大学DNA存储团队提出DNA存储基于隐藏参考序列的读出新方法
2026-01-14
微电子学院第四期学生骨干芯火班联合太雷班开展“理解中国”红色基因读书会
2026-01-12
微电子学院2025年度学生党支部书记述职评议会暨工作沙龙
2026-01-09
微电子学院学生党支部联合举办“学贺词凝心铸魂 启新程奋楫扬帆”主题党日活动
2026-01-07
【喜报】我院研究生李祖宇荣获“北洋体育风采奖”
2025-12-30
共青团天津大学微电子学院第二次代表大会顺利召开
2025-12-30
【二五逐光 芯向微芒】这场迎新晚会后,我产生了“戒断反应”
2025-12-29
【喜报】我院新增两门国家级一流本科课程

